Uma vez com o banco de dados importados, podemos prosseguir com a realização da metanálise. Utilizaremos o pacote meta para rodar essas análises. O meta é uma extensão para o R com funções implementadas que permite a realização de metanálises. Explicamos como instalar o pacote meta anteriormente; contudo, para prosseguir é necessário habilitá-lo. Assim, no quadrante inferior direito do R, em packages, clique em meta.
Para realizar uma metanálise de desfechos categóricos, utilizaremos o comando metabin. Os argumentos do comando, assim como informações adicionais, podem ser encontradas no material de ajuda do pacote, para isso basta digitar no console help (metabin) .
Os argumentos básicos para o comando metabin são a amostra e o número de eventos dos grupos intervenção e controle, e os nomes dos estudos (para essas informações são utilizados os rótulos de cada coluna correspondente no banco), além do nome do banco de dados a ser utilizado, com esses argumentos entre parênteses e separados por vírgula, na ordem a seguir:
metabin (eventos_grupo_intervenção, amostra_grupo intervenção, eventos_grupo_controle, amostra_grupo_controle, estudos, data=nome_do_banco)
Criaremos uma variável como esse resultado da metanálise criada a partir do metabin, daremos o nome de “metamacro”. Assim, a linha de comando será:
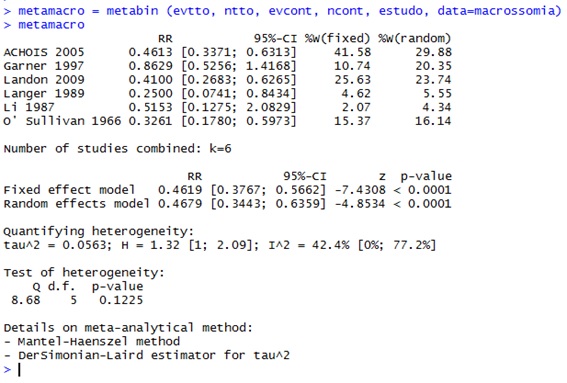
metamacro = metabin (evtto, ntto, evcont, ncont, estudo, data=macrossomia)
Aparentemente não aconteceu nada, mas o R salvou o resultado da metanálise dentro da variável metamacro. Ao digitar metamacro no console, o software nos mostrará os resultados.
Temos então o sumário dos resultados da metanálise. Na primeira parte temos cada um dos estudos, com seu risco relativo, intervalos de confiança e o seu peso nas análises tanto pelo modelo de efeitos fixos quanto pelo modelo de efeitos aleatórios. A seguir temos a medida sumária (RR e IC95%) da metanálise, apresentada para modelo de efeitos fixos e aleatórios, e seus respetivos valores P. Depois temos medidas de heterogeneidade da metanálise (valores para inconsistência e teste Q).
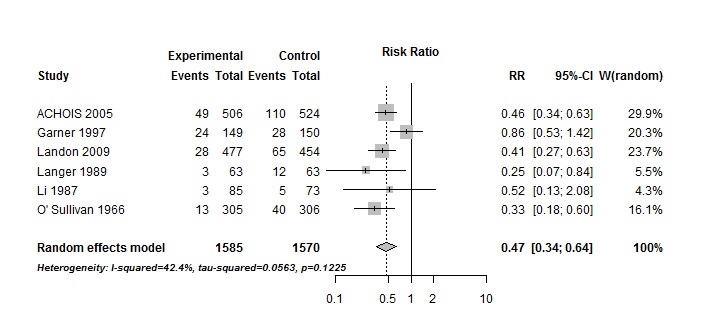
Para realizar o forest plot da metanálise utiliza-se o comando forest. Ao digitar ” forest (nome da metanálise) ”, o R irá criar um forest plot da metanálise. Nesse caso digite forest (metamacro) no console. Caso queira omitir no gráfico o resultado do modelo contando efeitos aleatórios, defina o argumento comb.fixed como falso, digitando no console a seguinte linha de comando:
forest (metamacro, comb.fixed=FALSE)
Assim temos o gráfico sumário da metanálise realizada, com o resultado combinado apenas com o efeito fixo. O gráfico é bastante customizável, parâmetros adicionais para o comando forest podem ser consultados através do arquivo de ajuda, digitando help (forest) no console.
A conclusão dos dados nessa metanálise é que o risco é menor da ocorrência de macrossomia no grupo intervenção (RR=0,47). Podemos dizer que o tratamento para diabetes gestacional reduziu em 53% a incidência de neonatos com macrossomia, comparado com a ausência de tratamento; a inconsistência pode ser considerada moderada (I2=42,4%).
Esperamos que esse tutorial tenha sido útil para seu aprendizado. Sugerimos prosseguir os estudos com a leitura do manual introdutório do R e com a documentação do pacote meta, que possui os comandos, argumentos e exemplos para realização de metanálise em R.
Caso deseje aprofundar seus estudos, possuímos curso presencial específico de metanálise em R, para maiores informações, acesse esse link.


Adoraria participar do curso, mas infelizmente moro fora do Brasil. Se for possivel fazer online via skype no dia do curso adoraria!!
Mais uma vez muito obrigada!! Parabens pelo blog e organização dos cursos!!
O pouco que domino aprendi no Stata, mas estou me desafiando a aprender a linguagem R pelo fato dele ser gratuito. Esse post me ajudou muito e pretendo fazer o curso com vocês tão logo seja possível. Uma dúvida: todos os cursos são com R ou tem cursos com Stata?
Olá Hugo,
O workshop de metanalise em R é dado em R (+RStudio) com o pacote meta (eventualmente metafor e mada). O de MTC é dado em openbugs + R, pacotes meta e gemtc. O curso introdutótio utiliza o RevMan e o de testes diagnósticos utiliza o metaDisc.
Nenhum curso utiliza o stata e nenhum curso exige conhecimento prévio do software. A escolha do R em vez do stata pesa pelo fato de ser gratuito e de fácil disponibilidade, mas o stata cumpre basicamente o mesmo objetivo.
Abraço